トピックス
HTLV-1プロウイルスゲノムにおける新規エンハンサー領域の特定と機能解析
ポイント
- ヒトT細胞白血病ウイルス1型(HTLV-1)は、細胞に感染するとウイルスゲノムDNAが宿主細胞ゲノムDNAに組み込まれるため、感染者からウイルスを排除することが極めて難しく、慢性持続感染を引き起こします。本研究ではその慢性持続感染成立に重要なウイルス内の新たな領域(エンハンサー領域※1)を発見しました。
- 感染者の体内で起きている現象を調べるために、実際の感染者検体を用いて最先端のシングルセル解析技術を駆使して研究を進めた結果、今回の新しい発見につながりました。
- これらの発見は、HTLV-1感染の持続性や病原性を理解する上で重要な知見であり、HTLV-1慢性持続感染を阻止する新たな治療標的の創出へと繋がる研究成果です。
研究概要
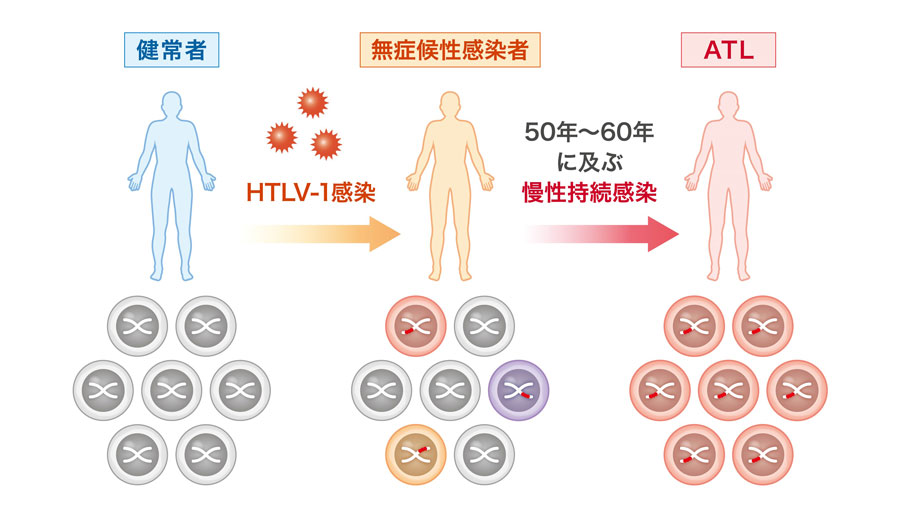
HTLV-1はヒトに病原性を示すレトロウイルスであり、ヒトの免疫システムで中心的働きをするCD4+T細胞を主な感染標的細胞とします。このウイルスは宿主細胞内に侵入したのち、細胞のDNAゲノムにウイルスDNAを挿入しプロウイルスとして維持されます。そのため、ひとたび感染が成立すると慢性持続感染状態となりますが、感染者の大部分は関連疾患を発症すること無く、その生涯を無症候性感染者として過ごします。その一方で2-5%の感染者は約60年という非常に長い潜伏期間を経て、成人T細胞白血病(ATL)を発症します(図1)。ATLは極めて予後不良な白血病であることが知られています。
ATL細胞の宿主ゲノムDNAにはプロウイルスDNAが存在します。HTLV-1に感染していなければCD4+T細胞のがんは極めてまれなため、プロウイルスの存在が細胞がん化を誘導すると考えられています。したがって、組み込まれたプロウイルスについて多くの研究が進められてきましたが、ウイルス発見から40年以上、今回特定されたウイルスエンハンサーの存在は知られていませんでした。
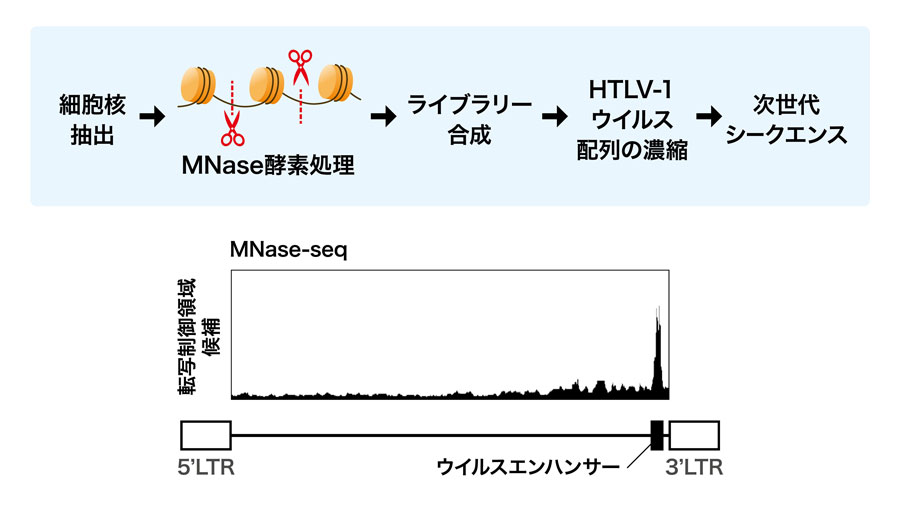
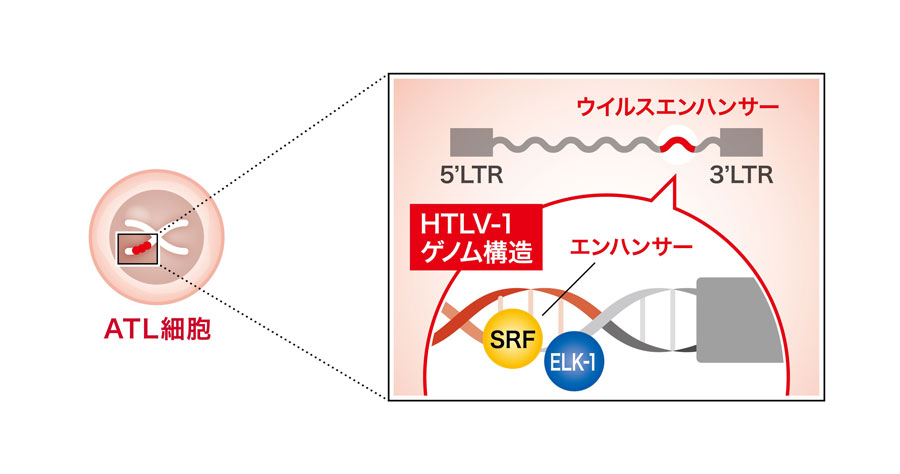
私たちは、九州の医療機関と連携し、実際の感染者検体を用いて、高感度ウイルス配列解析手法を用いたMNase-seqを実施することで、HTLV-1プロウイルス内の転写制御領域について網羅的に調べました。その結果、ウイルスゲノム内にこれまで未特定であった転写制御領域(エンハンサー領域)を新たに特定しました(図2)。加えてウイルスがどのような仕組みでエンハンサー活性を獲得するかについて調べた結果、SRFとELK-1という2つの宿主細胞因子の関与が明らかとなりました(図3)。
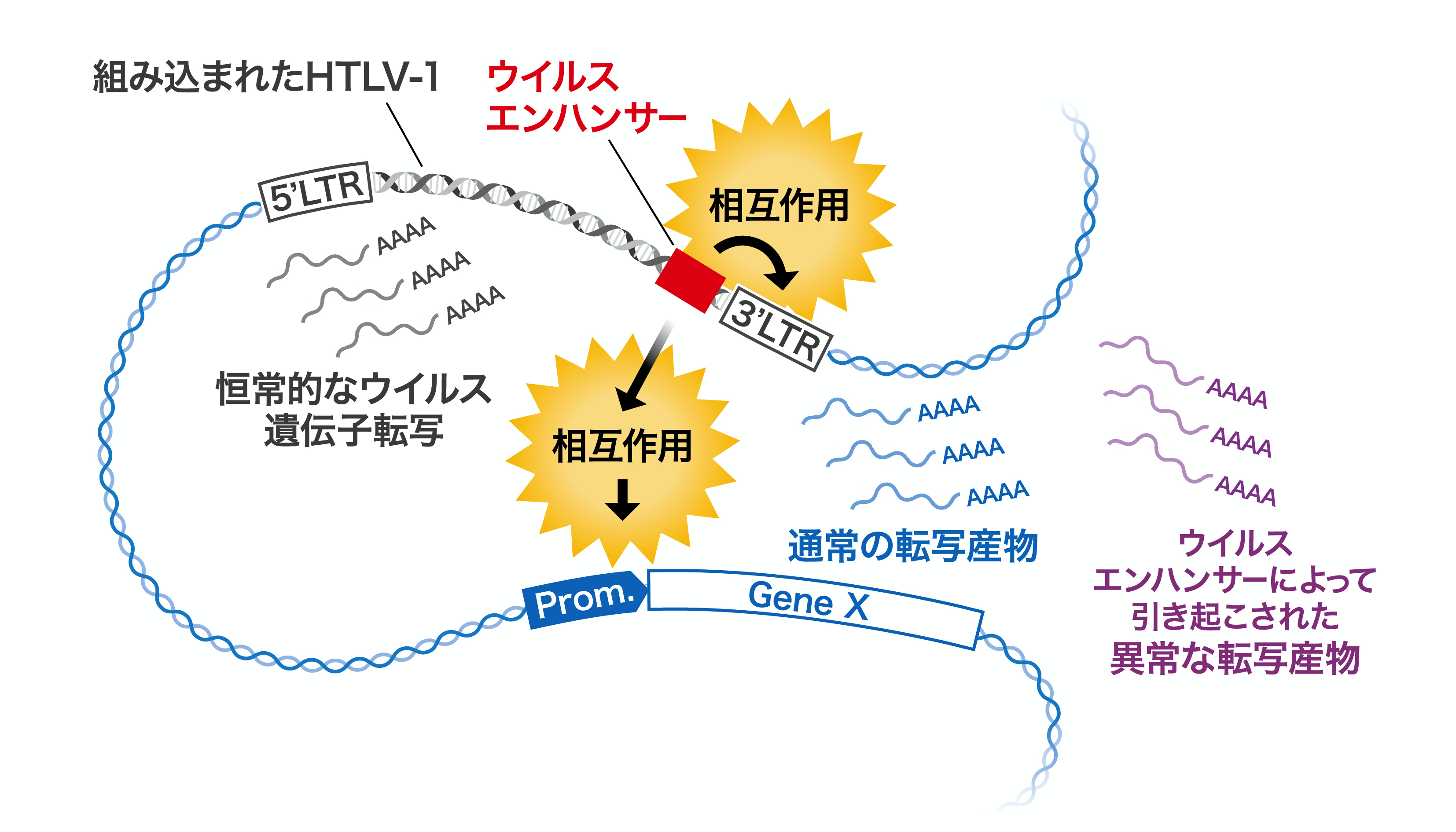
また、ATL細胞ゲノムに組み込まれたHTLV-1プロウイルス周辺で、宿主細胞の遺伝子発現が活性化されることが知られており、このウイルスによる白血病発症の一因となることが報告されています。本研究において、今回特定されたHTLV-1ウイルスエンハンサーはウイルス遺伝子の発現維持に貢献するだけでなく、組み込み部位周辺の細胞側遺伝子発現にも働きかけ、ATL細胞における宿主遺伝子転写異常の一因となることが示唆されました(図4)。
今回の研究成果は、次世代シークエンサーやシングルセル解析などの先端的研究手法で実際の患者検体を高精度に解析することで、従来の手法では見つけることができなかった、HTLV-1プロウイルス内新規エンハンサー領域の存在を明らかにしたものです。本研究成果はHTLV-1感染の持続性や病原性の仕組みの一端を明らかにし、難治性慢性ウイルス感染症であるHTLV-1感染症の問題克服へ向け、一歩前進する重要な知見と考えられます。
論文情報
- 論文名:
- Identification and characterization of a novel enhancer in the HTLV-1 proviral genome
- 著者:
- Misaki Matsuo, Takaharu Ueno#, Kazuaki Monde#, Kenji Sugata#, Benjy Jek Yang Tan, Akhinur Rahman, Paola Miyazato, Kyosuke Uchiyama, Saiful Islam, Hiroo Katsuya, Shinsuke Nakajima, Masahito Tokunaga, Kisato Nosaka, Hiroyuki Hata, Atae Utsunomiya, Jun-ichi Fujisawa, Yorifumi Satou*
- 掲載誌:
- Nature Communications 2022
